吃与活
科学地吃,健康地活王传超,李辉
复旦大学 生命科学学院 现代人类学教育部重点实验室
原载于Investigative Genetics, 2013, 4(1):11, 王传超 译。
摘要:东亚人群有着极其丰富的遗传、体质、文化和语言多样性,但这些多样性的具体分布状况以及相互间的渊源关系仍有待进一步解析。随着东亚及其周边人群的分子人类学数据的不断积累,尤其是父系Y染色体研究的一系列进展,使得东亚人群的多样性结构逐渐明晰。现有的Y染色体数据揭示,现代人出非洲后由东南亚经多次迁徙进入东亚。在旧石器时代,现代人最初定居东亚之后,紧接着不断北迁,这奠定了东亚遗传结构的基础。随后,来自中亚的移民与东亚人群的基因交流增大了东亚的南北人群间遗传距离。语言、农业、军事与社会地位等文化因素同样影响着东亚的遗传结构。将Y染色体与家谱文献相结合也为遗传学研究人类古代历史提供了可能。
关键词:东亚人群,Y染色体,迁徙,遗传结构
东亚是亚洲的一部分,面积广阔、风光旖旎。东亚有着占世界22%的人口,主要分为四种体质类型:新石器时代东亚人,即蒙古人种;旧石器时代大洋洲人,即澳大利亚人种;旧石器时代东南亚人,即尼格利陀人种;还有欧洲人即高加索人种。东亚有阿尔泰、南亚、南岛、侗傣、苗瑶、汉藏和印欧等七个语系的200多种语言。这使得东亚成为世界上研究人类进化、遗传多样性和基因与文化相互作用的最重要区域之一[1]。
在过去数年中,分子人类学的研究者们通过使用常染色和X染色体、父系Y染色体、母系线粒体等等各类遗传标记体系来解析东亚人群的遗传多样性。常染色体和X染色体遗传自父母双方,会被重组所打乱,而Y染色体上主干的非重组区呈严格父系遗传,并且Y染色体的“有效群体大小”理论上至多为常染色体的四分之一,X染色体的三分之一,对漂变非常敏感,容易形成群体特异性多态标记,从而包含更多的关于群体历史的信息。Y染色体的这些特点使其成为研究人类进化和迁徙最强有力的工具之一[2,3]。
Y染色体进入人们的视野,开始于其在追溯现代人起源上的应用。自上世纪90年代以来,人类学界争论最激烈的话题,是东亚地区现代人的起源问题。由于东亚出土了大量的古人类化石,一些人类学家认为东亚地区的人类是本土连续进化的,支持全球现代人的多地区起源。然而,1999年宿兵等人[4]采用Y染色体非重组区的19个SNP来研究东亚人群,得出东亚地区现代人起源于非洲,并由南方进入东亚,而后向北方迁。随后,2000年柯越海等人[5]的对东亚地区12127份男性随机样本的Y染色体进行SNP分型研究。Y染色体突变M168被认为约是7.4万年前现代人走出非洲时所产生的突变,其原始型仅出现在东非人群中,除非洲以外的人群都是突变型。柯越海等人的研究虽然没有直接检测M168这个突变,但他们检测了M89、M130和YAP这三个M168 下游的突变。结果显示这一万多份样品无一例外都带有M89、M130和YAP三种突变之一,也就是说都是M168突变型。尽管现在来看,东亚现代人或也与一些古人种有基因交流,但从父系角度看,现存的东亚人群都是走出非洲的后裔,这是支持现代人非洲单一起源的强有力的遗传学证据。下一问题就要回答早期现代人是如何迁徙来到东亚的。
人群的迁徙和分布与气候的变迁有着密切的关系,为便于从不同角度探索和认识人群演变规律,我们这里介绍一些近10万年来的气象学的材料。在距今约11万-1万之间,也就是考古学上的旧石器时代到中石器时代,地球处于末次冰期[9],那段时间,海平面远低于现在,许多现在的岛屿与大陆相连,成为了人类迁徙的重要通道。距今2.65万年到1.9-2万年是末次冰盛期,是末次冰期中气候最寒冷、冰川规模最大的时期,亚洲的绝大部分、北欧和北美都被冰雪覆盖,人类的生存空间也随冰川蔓延而逐渐缩小。大约1.5万年前,气温开始转暖,冰川也开始退却,现代人才迎来了迁徙的黄金时期[10,11]。
本文中,我们主要应用Y染色体的数据来分析东亚人群的迁徙历史,并探讨现代人最初定居东亚及其后的迁徙和扩张模式、微进化历程等。
北方路线还是南方路线?
现阶段比较一致的看法是东亚的欧洲人种类型来自西北[10, 12]、澳大利亚人和尼格利陀人来自东南[4,10],最具争议的还是蒙古利亚人来自哪里。有三种可能的模式:1)蒙古利亚人由北向南迁徙,与东南亚和中国南方的尼格利陀和澳大利亚人种混合;2)蒙古利亚人来自南方;3)北方人群来自北方,南方人群来自南方,自1万多年前的晚更新世以来,蒙古利亚人在南北方共同进化[13]。Y染色体是解决这一争议的有力工具。
Y染色体可以分为20种主干单倍群,编号从A到T(P可能不存在),其中O-M175, C-M130, D-M174和N-M231是东亚四个主要单倍群,约占到东亚全部男性的93%(图1)。其他单倍群,例如E-SRY4064, G-M201, H-M69, I-M170, J-P209, L-M20, Q-M242, R-M207和T-M70仅占到东亚男性的7% [12]。

图1. Y 染色体总单倍群C、D、N和O在东亚的地理分布。
O-M175是东亚最大的单倍群,约75%的中国人以及超过50%的日本人都可归到这一类型下,因此有理由认为它代表着蒙古利亚人。O-M175分出三个主要的下游单倍群O1a-M119、O2-M268以及O3-M122,这三个单倍群约占到东亚男性的60% [14,15]。O1a-M119在中国东南沿海、侗傣族群、台湾原住民中集中分布 [16]。O2-M268约在汉族中占5%以上 [14],O2a1-M95是O2下的主要支系,在华南、南方少数民族、中南半岛及印度门哒人群中分布较多 [16,17]。O2b-M176是O2下的另一支系,最主要集中于朝鲜半岛、朝鲜族和日本弥生人,越南人和汉族中也有少量分布 [18,19]。O3-M122是中国最常见的单倍群,遍及整个东亚和东南亚,占汉族50-60%左右。O3a1c-002611、O3a2c1-M134和O3a2c1a-M117是O3下的三个主要支系,各占到汉族的12-17%左右。O3a2c1a-M117在藏缅族群中也有较多分布。O3下的另一支系O3a2b-M7在苗瑶和孟高棉人群中高频出现,但在汉族中却不足5% [14,15]。
宿兵等[4]在亚洲大范围群体样本中对包括M119、M95和M122在内的19个Y染色体SNP位点以及三个STR位点进行了检测。在随后的主成分分析中,北方人群紧密聚在一起,且都被包含在南方人群的聚类簇之内,南方人群比北方人群多样性更高。他们认为北方人群来自于旧石器时代定居南方的南方人群。他们还使用STR位点的一步突变模式和0.18%突变率估算O3-M122这一单倍群的时间为1.8-6.0万年,这一时间可能反映的是最初定居东亚的瓶颈时期。2005年,石宏等[15]对东亚多个群体的2000多个O3样本进行了更系统的研究,他们的研究也发现南方群体中的O3-M122的多样性高于北方,支持O3-M122的南方起源。他们进一步使用均方差(ASD)方法和STR的进化突变率(0.00069每位点每25年)[20,21]估算O3支系北迁的时间为2.5-3.0万年。最近,蔡晓云等[22]对东南亚的孟高棉和苗瑶族群中的O3a2b-M7和O3a2c1a-M117进行了系统研究,揭示其在1.9万年前末次盛冰期时候经由东南亚进入东亚的单向瓶颈扩散[22]。O3下的另一主要支系O3a1c-002611的STR位点多样性也与其他兄弟支系一样有着大体上的自南向北递减的趋势[23]。总体来看,绝大多数证据都支持Y染色体单倍群O3-M122经由南方路线进入东亚并逐渐向北扩散(图2)。
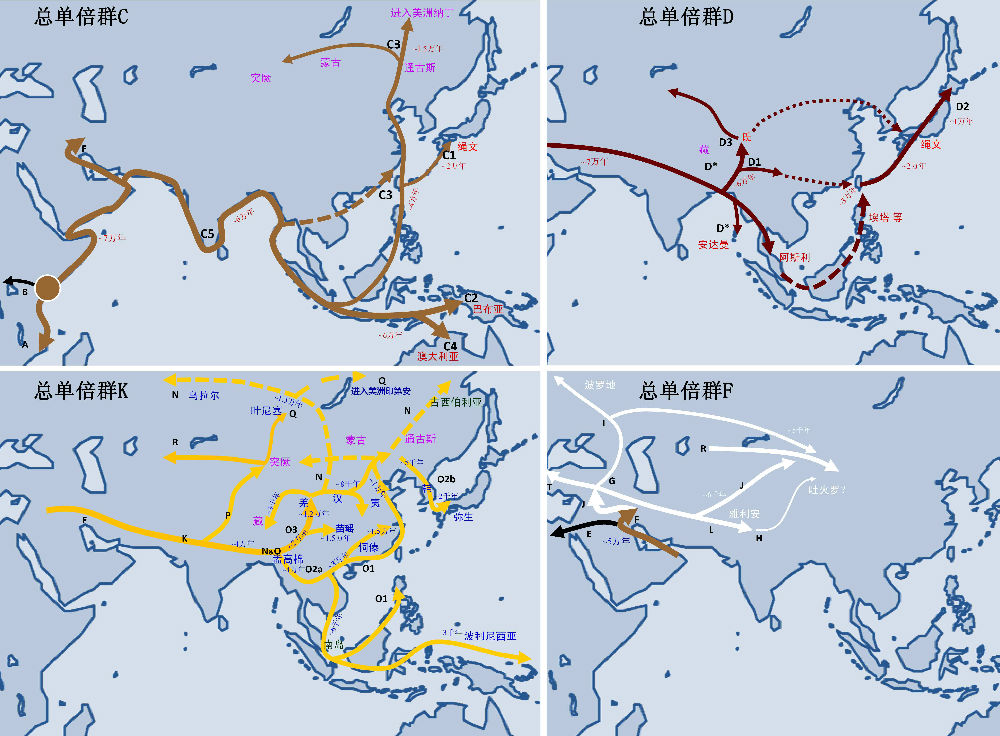
图2. Y 染色体单倍群C、D、N和O在东亚的迁徙。虚线表示另外可能的迁徙路线。
东亚最早的定居者
东亚的特征单倍群O-M175的产生时间,由足够多的STR的位点估算很可能不超过3万年,因此单倍群O很可能根本不是东亚最早的定居者。单倍群C-M130的人群却极可能是最早到达东亚的人群。单倍群C从阿拉伯半岛南部、巴基斯坦、印度、斯里兰卡、东南亚、东亚、大洋洲到美洲都有分布,尤其在远东和大洋洲高频分布,但在撒哈拉以南的非洲没有被发现(图1)。C下游的分支,例如C1-M8、C2-M38、C3-M217、C4-M347、C5-M356和C6-P55,都有着区域特异性分布[24]。C3-M217是分布最广的支系,在蒙古和西伯利亚群体中最高频出现。单倍群C1仅在日本人和琉球人中出现,但频率很低,还不足5%。单倍群C2出现在从印度尼西亚东部到波利尼西亚的太平洋岛屿人群,尤其是在波利尼西亚的一些群体中,且由于连续的奠基者效应和遗传漂变而成为了上述地方的特征单倍群[19,25]。C4几乎仅局限在大洋洲的澳大利亚原住民中。C5在印度及其周边的巴基斯坦和尼泊尔等地低频出现[26,27]。C6则仅出现在新几内亚高地上[28]。单倍群C的分布模式说明了这个单倍群很可能是在亚洲大陆起源,且那时还没到达东南亚。
为更清楚说明单倍群C的源流,钟华等[24]对取自东亚和东南亚140多个群体的465个单倍群C的样本检测了C内部的12个SNP和8个STR位点。他们发现C3的STR多样性最高出现在东南亚,且呈自南向北、自东向西递减的趋势,ASD方法估算时间落在3.2-4.2万年间,这表明了旧石器时代C3是沿海岸线逐渐向北扩张的(图2)。单倍群C很可能在6万年前就已到达东南亚和澳大利亚,比其向北扩散的时间要早得多,这也就是说单倍群C在蒙古利亚人(单倍群O)来之前的就已在东亚生活了数万年。经过如此长的时间,单倍群C的人群或已与蒙古利亚人有着不同的体质特征。因为现在单倍群C的人群多有着澳大利亚人的体质特征,例如澳大利亚原住民、巴布亚人和一些达罗毗荼人,所以我们认为单倍群C是由澳大利亚人体质特征的人带来的,他们达到远东的时间要早于其他现代人。北京周口店出土的1万年前的人骨就有着澳大利亚人的体质特点,或也支持澳大利亚人是东亚最早定居者。
东亚的黑人遗存
最具神秘色彩的是Y染色体单倍群D的迁徙历史,迄今为止我们仍对此知之甚少。单倍群D是从非洲的DE-M1(YAP插入)单倍群衍生出来的,很可能与矮黑体质的尼格利陀人相关联。单倍群E是D的兄弟支系,E随着高黑人西迁非洲,D则可能由矮黑人东迁带到东亚(图3)。
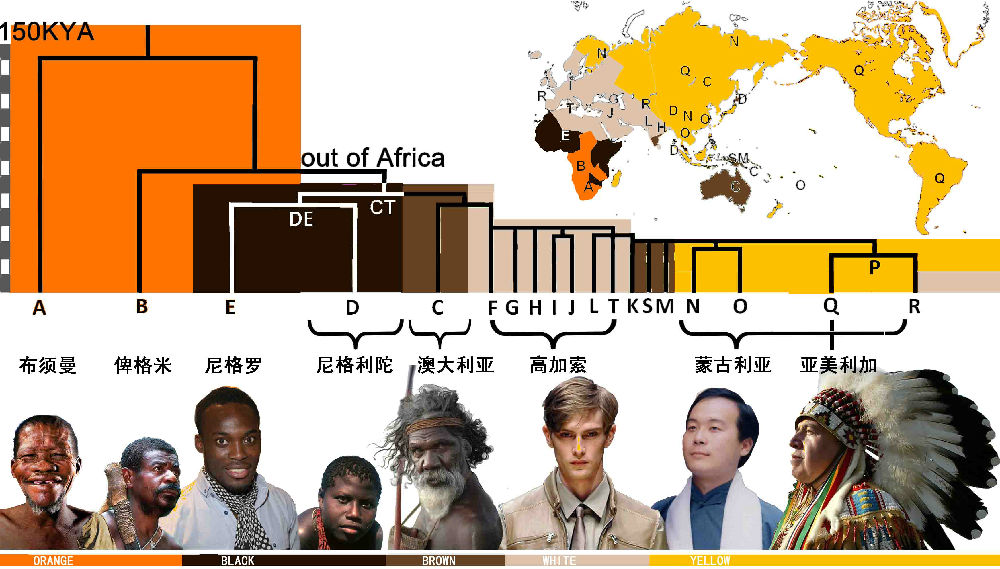
图3. Y染色体谱系的主干以及其与不同体质特征的现代人可能联系。
单倍群D-M174在安达曼尼格利陀人、北部藏缅群体和日本的阿伊努人中高频分布,在其他东亚、东南亚和中亚群体也有低频分布(图1)[17,19,29,30]。D下分D1-M15、D2-M55和D3-P99三个主要支系,还有许多未明确定位的小支系。D1在藏族、羌语支和彝语支人群中广泛分布,在东亚其他群体中也有低频分布[31,32]。D2仅分布于日本,占日本40%以上,是上古绳文人的主要成分。D3在青藏高原东部(康区)、白马人及纳西族等群体中高频 [31]。D*多在安达曼群岛被发现[30],且已被隔离了至少2万年。其他一些被包含在D*中的小支系也多分布于西藏周边藏缅语人群、东南亚人群,阿尔泰人中也有少量来源不明的D*。这些D*的内部谱系需要详细调查分析。单倍群D高频人群的肤色大多较深,包括安达曼人、一些藏缅和孟高棉人等。阿伊努人肤色变白可能是为了吸收更多紫外线以适应高纬度地区生存。
对于单倍群D的起源,Chandrasekar等认为CT-M168在南亚分出了YAP插入和D-M174突变,因为他们在印度东北一些族群中发现有YAP插入而在安达曼群岛上检测到了M174突变 [33]。这样来看,同样带有YAP插入的E单倍群也很可能是亚洲起源,但没有证据进一步支持。如果单倍群D诞生于非洲,那非常有趣的是它是如何随着总单倍群CF的群体来到东亚的?
另一不可思议的是单倍群D是如何由东亚的西南角一路去到日本的。它可能通过东亚大陆北上,还可能是经由巽他大陆,但穿过东亚大陆似乎更近。石宏等人推论单倍群D北上扩张到中国西部的时间约在6万年前(ASD方法),要早于东亚其他主要支系的迁徙。随后,这一先头部队可能通过北向路线经由朝鲜半岛到达日本,或者通过南向路线经由台湾和琉球所形成的大陆桥到达日本,这一过程中他们可能与澳大利亚人相遇过。后来单倍群O的北上以及新石器时代汉族扩张,单倍群D的主体人群可能就被挤出了中国东部 [31]。但是无论是遗传学上还是考古学上都没有任何证据表明D2或尼格利陀人曾到过中国大陆东部。相反,从马来半岛到波利尼西亚的巽他大陆至今仍有大量的尼格利陀人。尼格利陀人或许在旧石器晚期占据了整个巽他大陆。那么,这些人群可能直接从菲律宾到台湾和琉球。唯一难以解释的是在菲律宾的尼格利陀人中从未发现过D的存在,他们的父系或许已在约1.8万年前(BATWING方法)被来自于巴布亚岛的C2和K的扩张所取代[34],当然也可能被非常晚近时期来自于东亚大陆的单倍群O所替换[35]。因为相关数据的不足,东亚的黑人遗存-单倍群D的源流还远未揭开。
往来西北的迁徙
单倍群O的兄弟支系是单倍群N-M231,单倍群N在欧亚大陆北部,尤其是包括芬兰、乌戈尔、萨摩耶德和尤卡吉尔等分支的乌拉尔语人群、以及阿尔泰语人群和爱斯基摩人中高频分布,它还低频出现在东亚内陆(图1)[29,36]。对于单倍群N 的详细分析显示,N 在东欧的高频是缘于很晚近的迁徙,这次迁徙约从1.2万到 1.4万年前(ASD方法)开始,由内亚/南西伯利亚出发,走一条逆时针的北部路线 [36]。N的下游分支N1a-M128低频分布于中国北部一些群体,例如满族、锡伯族、鄂温克族和朝鲜族,以及中亚的一些突厥语族群中。另一分支N1b-P43在北部的萨莫耶德人中广泛分布,也在一些乌拉尔和阿尔泰人群中低频或中频分布,N1b大约在6-8年前诞生于西伯利亚[37,38]。频率最高的下游单倍群N1c-Tat,可能在1.4万年前起源于中国西部地区,然后在西伯利亚经历多次瓶颈效应,最后扩散到东欧和北欧 [36]。这些研究把单倍群N的起源追溯到中国西南或东南亚,单倍群N的人群艰苦跋涉由东南亚穿越大陆一直到北欧,谱写了壮丽的迁徙史诗。
单倍群N的迁徙史为东亚人群南方起源提供了又一项强有力的证据。然而仍有一些研究在质疑南方起源。Karafet等对来自东亚和中亚地区的25个群体的1300多份样本进行Y染色体分型研究,他们发现各单倍群间的两两差异在东亚南部是非常小的,且东亚南北群体之间并未发现遗传分化 [29]。薛雅丽等[39]使用贝叶斯全似然法来分析取自中国、蒙古、韩国和日本的27个群体近1000份样本45个Y染色体SNP和16个STR位点。他们发现东亚北方群体的Y染色体的STR多样性要高于南方,北方群体的扩散要早于南方群体[39]。但随后石宏指出Karafet所观察到的北方群体的高多样性应是由近期的人群混合造成的,薛雅丽等分析结果也存在这一问题,蒙古、维吾尔和满族的基因多样性高应是由他们与西方人群以及汉族大规模混合的结果。另外,薛雅丽等所选取的南方群体代表性不够,长期地理隔离所造成的群体内部的瓶颈效应或对基因多样性的估算有较大影响 [31]。
后续的争论就集中在如何辨析中亚和欧亚西部人群对东亚的基因贡献。钟华等[13]对117个群体的近4000份样本的Y染色体进行高分辨率的分型判断来试图阐明这一问题。在钟华等的研究中,单倍群O-M175、C-M130、D-M174和N-M231仍显出了南方路线较大基因贡献。然而,与中亚和欧亚西部相关的单倍群,例如单倍群R-M207和Q-M242,多在东亚西北地区出现,且它们的频率自西向东有递减的趋势。另外,单倍群R-M207和Q-M242的Y染色体STR多样性也提示了北方路线存在的可能性,也就是1.8万年前由中亚到北亚的迁徙和3千年前沿丝绸之路的人群混合。
母语还是父语?
人群的遗传模式常会被其居住习俗和生存方式等社会文化因素所影响。东亚人群的Y染色体可以很好地反映这些文化烙印,例如从父居的群体之间应表现出族群关系与父系的Y染色体较强的关联性而不是与母系的线粒体。东亚的语言确实与父系的Y染色体[15,22,24,40,41]和整个基因组多样性[42]有较强的对应关系,但不与母系线粒体相关。例如,Y染色体单倍群O3-M134与汉藏语系人群相关[15,40],O2-M95与南亚语系相关[41]。而且,语言学家们所提出的语系之间的系统发生关系也可反映在Y染色体上但不与全基因组多样性相关,例如,苗瑶和南亚语系间的近缘性由单倍群O3-M7所反映出来[22],侗傣语系和南岛语系间的近缘性由O1-M119所反映[43]。
另一有趣的话题是关于语言扩张和Y染色体的分布模式。现代语言在扩张过程中是否也经历了一系列的奠基者效应?Atkinson发现音素(元音、辅音和声调)的多样性有着出非洲的递减趋势,因而推论现代语言也是源出非洲 [44]。但是这一论断并未得到语言学界的广泛认可。王传超等指出Atkinson的结论仅在音素多样性被简单分为3-5类之后才成立,而应用没有简化的音素原始数值进行分析却得出了由亚洲中部向外的多样性递减趋势[45],当然这不是关于语言源流的最终结论[46]。音素多样性的分布模式反映的或许不是现代人的最初起源过程,而是现代人在亚洲中南部的二次扩张。Y染色体的数据也支持亚洲扩张。Y染色体谱系树的根部在非洲,但仅是单倍群A和B是非洲土著类型,这两个单倍群或许从未离开非洲。其他单倍群都在CF和DE之下,均源自一个5-7万年前出非洲的古老突变M168。他们或在3-4万年前从西亚扩张开来,并衍生出了由C到T的所有单倍群 [3]。因此,在非洲高频出现的单倍群E很可能来自于亚洲扩张后的回流。在非洲,单倍群A集中出现在科依桑人和撒哈拉人中,单倍群B则主要出现在俾格米人及刚果周围的其他群体中。非洲的主要群体,也就是班图人或尼日尔-刚果人,或许都是亚洲回流到非洲的。 单倍群DE-M1(YAP+)经由亚洲返回非洲已被一些研究者提出[47-49],但这推论也遭到了其他人质疑[50,51]。此外,在喀麦隆高频出现的R1-M173更明确地支持由亚洲向撒哈拉以南非洲的人群回迁[52]。
语言与Y染色体相关但不与线粒体相关,或许反映的是由从父居所引起的性别偏向性迁徙过程。从父居指得是夫妻婚后与丈夫的父母住在一起或住在他们附近。Forster等指出如果父母双方语言不同,那么多是父亲的语言在家庭中占主导地位 [53]。然而,因为全基因组多样性也与语系相关,语系形成之时父系和母系应该已完好保存下来。因此,线粒体与语言间的不相关或不能简单地用从父居来解释。有可能是因为狩猎或战争使得最初群体中女性的有效群体就大于男性,那么同一语言群体中线粒体受到遗传漂变得影响就小。当然其他解释,例如优选男性(下文具体介绍)、男性生育周期长、后代的数目以及突变率的不同等也都有可能。
其他的文化因素,例如农业、军事和社会地位等,也都有可能影响遗传模式。文化的传播模式有两种:一是人口流动驱动文化传播,二是单纯的文化传播,不涉及人群的流动[54]。举例而言,近东到欧洲的农业传播是否伴随着大量的新石器农民不断迁徙一直是争论的焦点 [55]。Chikhi等应用家系似然法去分析包含22个Y染色体SNP的大批量数据,他们发现近东的农业进入欧洲伴随着大量的基因流动,这支持了人口流动驱动文化传播的模式 [56]。另一个例子,文波等研究了28个汉族群体的Y染色体和线粒体的遗传结构,他们的结果显示南北方汉族有着相近的Y染色体结构,但是南北汉族在线粒体上却有着较大差别。或许是由于战乱或饥荒,大量的北方移民来到南方而改变了中国南方的遗传结构。他们认为汉族人口扩张和汉文化的南下是符合人口驱动的文化传播模式的,而且在这次扩张中男性占主体地位。
历史名人的Y染色体
人群扩张还可以与特殊的社会地位相联系起来,比如成吉思汗家族。成吉思汗(1162-1227)南征北战,建立了历史上疆域最辽阔的国家。他和他的父系亲属因其很高的政治地位而有不计其数的后代,这无疑增高了他们的Y染色体在群体中所占的频率。结果,可能是成吉思汗或者其近亲宗族的Y染色体类型(C3*xC3c,星簇)出现在了从太平洋一直延伸到里海广阔地域,占到了全世界男性的0.5% [57]。有趣的是,C3*星簇最高频地出现在哈萨克斯坦的克烈部 [58]。克烈部高频的C3*星簇难以归因于成吉思汗,成吉思汗家族的Y染色体类型或许不是星簇。无论如何,社会选择确实在C3*星簇的扩张中起到了重要作用。同样地,Y染色体单倍群C3c-M48被推断为清朝(1644-1912)满族皇室的类型,占到了东亚男性的3.3% [59]。
与研究成吉思汗的谱系一样,Y染色体可以用来追溯历史名人。人们的姓氏大多继承自父亲,而Y染色体是严格的父子相传的基因组片段。所以姓氏与Y染色体的遗传应该是平行的,有共同姓氏的男性可能有相同或相近的Y染色体类型。那么,结合家谱材料,通过研究历史人物现存后代的Y染色体,可以揭示历史人物之间的父系关系 [60]。王传超等用Y染色体分型比对的方法确认了若干有1800多年历史、延续70-100代的大跨度家系,这些家系宣称是魏武帝曹操后裔。曹操后裔的Y染色体类型为O2*-M268,与西汉丞相曹参后裔的Y染色体O3-002611并不一致。所以,曹操一直自称的源自曹参的贵族血统并不被遗传学所支持 [61]。
将遗传学应用到古代史研究将会不断增多。举例而言,在家谱学上,同一姓氏的不同家族可以通过遗传学检测来填补谱牒材料的缺环 [62]。深度家系对于研究Y染色体的进化也有很大价值,例如薛雅丽等[63]通过测序相隔13代的两个体的Y染色体得出Y染色体上的碱基突变率为3.0x10-8每突变/每世代。更深度的家系将会是更准确估算突变率的更好的材料。
总结展望
Y染色体在解析东亚现代人源流史中起到了重要作用。尽管有许多问题仍有待探索,但史前迁徙过程的基本框架已经明晰了。占到东亚男性90%以上的C、D、N和O四个单倍群很可能起源于东南亚,随澳大利亚人、尼格利陀人和蒙古利亚人这三种不同体质特征的现代人经历了三次大的迁徙浪潮。欧亚中西部特征Y染色体单倍群E、G、H、I、J、L、Q、R和T在中国西北的分布模式反映出了来自西方的近期基因交流和可能的北部路线的影响,这些单倍群自西向东的递减趋势也可以清晰地观察到。
然而,现阶段东亚的Y染色体研究遇到了两个瓶颈。一是东亚特异单倍群O-M175的解析度太低。虽然,单倍群O人口众多,但O下的位点却比R和E都少。例如,002611、M134和M117这三个位点代表了东亚近2.6亿人,但没有更下游的位点可以用来更精细解析这些群体的遗传结构。另一个瓶颈是支系和群体分化时间的估算。现在绝大部分的时间估算用的是Y染色体的STR位点,尽管这在理论上说得通,但对于最恰当的STR估算时间的方式还一直有争议。尤其值得提出的是这里有两种经常用到的Y染色体STR突变率,即进化突变率 [20,21]和家系突变率[64]。如何选用这两种突变率争议很大,因两者估算出的时间甚至可相差3倍。而且STR位点的相似性及多变性也使得时间估算的准确度大打折扣。因此,上文提到一些时间点也仅仅是作为某些单倍群或人群分化的粗略参考。
二代测序技术的不断发展使得全测序大样本量和深度家系的Y染色体成为可能。例如,千人基因组计划在其低覆盖项目中已经以1.83的平均深度测序了77个男性的Y染色体,15.23的深度测序了两个连续三代的男性家系 [65]。更进一步的深度测序将既可以细化Y染色体谱系树又可以为进化研究提供较精确的生物钟校准。
致谢
本项目得到了国家自然科学基金(31071098, 91131002),国家优秀青年科学基金(31222030),上海市启明星计划(12QA1400300),教育部重点项目(311016),上海市教委科技创新重点项目(11zz04),上海市人才发展资金(2010001) ,复旦大学文科科研推进计划的资助,特此致谢!
参考文献
1. Cavalli-Sforza LL: (1998) The Chinese human genome diversity project. Proc. Natl Acad. Sci. USA, 95: 11501–11503.
2. Jobling M A, Tyler-Smith C. (1995 ) Father and sons: the Y chromosome and human evolution. Trends Genet, 11: 449–456.
3. Underhill P A, Shen P, Lin A A, et al. (2000) Y chromosome sequence variation and the history of human populations. Nat. Genet, 26: 358–361.
4. Su, B. Xiao J, Underhill P, et al. (1999 ) Y-chromosome evidence for a northward migration of modern human into East Asia during the last ice age. Am J Hum Genet, 65: 1718–1724.
5. Ke Y, Su B, Song X, et al. (2001) African origin of modern humans in East Asia: a tale of 12,000 Y chromosomes. Science, 292:1151-1153.
6. Green R E, Krause J, Briggs A W, et al. (2010) A draft sequence of the Neandertal genome. Science, 328:710-722.
7. Reich D, Green R E, Kircher M, et al. (2010) Genetic history of an archaic hominin group from Denisova Cave in Siberia. Nature 2010, 468:1053-1060.
8. Wang C C, Farina S E, Li H: (2012) Neanderthal DNA and Modern Human Origins. Quatern Int, doi:10.1016/j.quaint..02.027.
9. Shi Y F, Cui Z J, Li J J. (1989) Quaternary glacier in eastern China and the climate fluctuation. Beijing, Science Press.
10. Jobling M A, Hurles M, Tyler—Smith C. (2004) Human Evolutionary Genetics (Origins, Peoples and Disease). New York: Garland Science.
11. Clark P U, Dyke A S, Shakun J D, et al. (2009) The Last Glacial Maximum. Science, 325:710-714.
12. Zhong H, Shi H, Qi X B, et al. (2011) Extended Y chromosome investigation suggests postglacial migrations of modern humans into East Asia via the northern route. Mol Biol Evol, 28(1):717-727.
13. Piazza A: (1998) Towards a genetic history of China. Nature, 395: 636–639.
14. Yan S, Wang C C, Li H, et al. Genographic Consortium: (2011) An updated tree of Y-chromosome Haplogroup O and revised phylogenetic positions of mutations P164 and PK4. Eur J Hum Genet, 19(9):1013-1015.
15. Shi H, Dong Y L, Wen B, et al. (2005) Y-chromosome evidence of southern origin of the East Asian-specific haplogroup O3-M122. Am J Hum Genet 2005, 77(3):408-419.
16. Kayser M, Choi Y, van Oven M, et al. (2008) The impact of the Austronesian expansion: evidence from mtDNA and Y chromosome diversity in the Admiralty Islands of Melanesia. Mol Biol Evol 2008, 25(7):1362-1374.
17. Su B, Jin L, Underhill P, et al. (2000) Polynesian origins: insights from the Y chromosome. Proc Natl Acad Sci USA, 97(15):8225-8228.
18. Ding Q L, Wang C C, Farina S E, et al. (2011) Mapping Human Genetic Diversity on the Japanese Archipelago. Advances in Anthropology, 1(2): 19-25.
19. Hammer M F, Karafet T M, Park H, et al. (2006) Dual origins of the Japanese: common ground for hunter-gatherer and farmer Y chromosomes. J Hum Genet, 51:47–58.
20. Zhivotovsky LA. (2001) Estimating divergence time with the use of microsatellite genetic distances: impacts of population growth and gene flow. Mol Biol Evol, 18:700–709.
21. Zhivotovsky L A, Underhill P A, Cinniog? lu C, et al. (2004) The effective mutation rate at Y chromosome short tandem repeats, with application to human population-divergence time. Am J Hum Genet , 74:50–61
22. Cai X, Qin Z, Wen B, et al. (2011) Genographic Consortium: Human migration through bottlenecks from Southeast Asia into East Asia during Last Glacial Maximum revealed by Y chromosomes. PLoS One, 6(8):e24282.
23. Wang C C, Yan S, Qin Z D, et al. (2012) Late Neolithic expansion of ancient Chinese revealed by Y chromosome haplogroup O3a1c-002611. J Syst Evol, DOI: 10.1111/j.1759-6831.2012.00244.x
24. Zhong H, Shi H, Qi XB, et al. (2010) Global distribution of Y-chromosome haplogroup C reveals the prehistoric migration routes of African exodus and early settlement in East Asia. J Hum Genet, 55(7):428-435.
25. Kayser M, Brauer S, Cordaux R, et al. (2006) Melanesian and Asian origins of Polynesians: mtDNA and Y chromosome gradients across the Pacific. Mol Biol Evol 2006, 23: 2234–2244.
26. Sengupta S, Zhivotovsky L A, King R, et al. (2006) Polarity and temporality of high-resolution y-chromosome distributions in India identify both indigenous and exogenous expansions and reveal minor genetic influence of Central Asian pastoralists. Am J Hum Genet. 2006, 78: 202–221.
27. Gayden T, Cadenas A M, Regueiro M, et al. (2007) The Himalayas as a directional barrier to gene flow. Am J Hum Genet, 80: 884–894.
28. Karafet T M, Mendez F L, Meilerman M B, et al. (2008) New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree. Genome Res, 18:830–838.
29. Karafet T M, Xu L, Du R, et al. (2001) Paternal population history of East Asia: sources, patterns, and microevolutionary processes. Am J Hum Genet, 69(3):615-628.
30. Thangaraj K, Singh L, Reddy AG, Rao VR, Sehgal SC, Underhill PA, Pierson M, Frame IG, Hagelberg E: (2003) Genetic affinities of the Andaman Islanders, a vanishing human population. Curr Biol, 13(2):86-93.
31. Shi H, Zhong H, Peng Y, et al. (2008) Y chromosome evidence of earliest modern human settlement in East Asia and multiple origins of Tibetan and Japanese populations. BMC Biol, 6:45.
32. Wen B, Xie X, Gao S, et al. (2004) Analyses of genetic structure of Tibeto-Burman populations reveals sex-biased admixture in southern Tibeto-Burmans. Am J Hum Genet, 74(5):856-865.
33. Chandrasekar A, Saheb S Y, Gangopadyaya P, et al. (2007) YAP insertion signature in South Asia. Ann Hum Biol, 34:582-586.
34. Delfin F, Salvador J M, Calacal G C, et al. (2011) The Y-chromosome landscape of the Philippines: extensive heterogeneity and varying genetic affinities of Negrito and non-Negrito groups. Eur J Hum Genet, 19:224-230.
35. Scholes C, Siddle K, Ducourneau A, et al. (2011) Genetic diversity and evidence for population admixture in Batak Negritos from Palawan. Am J Phys Anthropol, 146:62-72.
36. Rootsi S, Zhivotovsky L A, Baldovic M, et al. (2007) A counter-clockwise northern route of the Y-chromosome haplogroup N from Southeast Asia towards Europe. Eur J Hum Genet, 15:204-211.
37. Derenko M, Malyarchuk B, Denisova G, et al. (2007) Y-chromosome haplogroup N dispersals from south Siberia to Europe. J Hum Genet, 52(9):763-770.
38. Mirabal S, Regueiro M, Cadenas A M, et al. (2009) Y-chromosome distribution within the geo-linguistic landscape of northwestern Russia. Eur J Hum Genet, 17(10):1260-1273.
39. Xue Y, Zerjal T, Bao W, et al. (2006) Male demography in East Asia: a north-south contrast in human population expansion times. Genetics, 172(4):2431-2439.
40. Wen B, Li H, Lu D, et al. (2004) Genetic evidence supports demic diffusion of Han culture. Nature, 431:302-5.
41. Kumar V, Reddy A N, Babu J P, et al. (2007)Y-chromosome evidence suggests a common paternal heritage of Austro-Asiatic populations. BMC Evol Biol, 7:47.
42. The HUGO Pan-Asian SNP Consortium.(2009) Mapping human genetic diversity in Asia. Science, 326: 1541.
43. Li H, Wen B, Chen S J, et al. (2008) Paternal genetic affinity between Western Austronesians and Daic populations. BMC Evol Biol, 8:146.
44. Atkinson Q D: (2011) Phonemic diversity supports a serial founder effect model of language expansion from Africa. Science, 332:346-349.
45. Wang C C, Ding Q L, Tao H, et al. (2012) Comment on "Phonemic diversity supports a serial founder effect model of language expansion from Africa". Science, 335:657.
46. Atkinson Q D. (2012) Response to Comment on “Phonemic Diversity Supports a Serial Founder Effect Model of Language Expansion from Africa”. Science, 335:657.
47. Hammer M F, Spurdle A B, Karafet T, et al. (1997) The geographic distribution of human Y chromosome variation. Genetics, 145:787–805.
48. Hammer M F, Karafet T, Rasanayagam A, et al. (1998) Out of Africa and back again: nested cladistic analysis of human Y chromosome variation. Mol Biol Evol, 15: 427–441.
49. Hammer M F, Karafet T M, Redd A J, et al. (2001) Hierarchical patterns of global human Y-chromosome diversity. Mol Biol Evol, 18:1189–1203.
50. Underhill P A, Passarino G, Lin A A, et al. (2001) The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations. Ann Hum Genet, 65:43–62.
51. Underhill P A, Roseman C C. (2001) The case for an African rather than an Asian origin of the human Y-chromosome YAP insertion. In: Jin L, Seielstad M, Xiao C (eds) Recent advances in human biology, vol. 8: genetic, linguistic and archaeological perspectives on human diversity in Southeast Asia. World Scientific Publishing, New Jersey, pp 43–56.
52. Cruciani F, Santolamazza P, Shen P D, et al. (2002) A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes. Am. J. Hum. Genet 70:1197–1214.
53. Forster P, Colin R. (2011) Mother Tongue and Y Chromosomes. Science, 333:1390-1391.
54. Cavalli-Sforza L L, Menozzi P, Piazza A. (1994) The History and Geography of Human Genes. Princeton: Princeton Univ. Press.
55. Sokal R, Oden N L, Wilson C. (1991) Genetic evidence for the spread of agriculture in Europe by demic diffusion. Nature, 351: 143–145.
56. Chikhi L, Nichols RA, Barbujani G, et al. (2002) Y genetic data support the Neolithic demic diffusion model. Proc Natl Acad Sci USA, 99:11008–11013.
57. Zerjal T, Xue Y, Bertorelle G, et al. (2003) The genetic legacy of the Mongols. Am J Hum Genet, 72: 717–721.
58. Abilev S, Malyarchuk B, Derenko M, et al. (2012) The Y-chromosome C3* star-cluster attributed to Genghis Khan's descendants is present at high frequency in the Kerey clan from Kazakhstan. Hum Biol, 84(1):79-89.
59. Xue Y, Zerjal T, Bao W, et al. (2010) Recent spread of a Y-chromosomal lineage in northern China and Mongolia. Am J Hum Genet 2005, 77:1112–1116.
60. Wang C C, Yan S, Li H:(2010) Surnames and the Y Chromosomes. Commun Contemp Anthropol, 4:e5/27-34.
61. Wang C, Yan S, Hou Z, et al. (2012) Present Y chromosomes reveal the ancestry of Emperor CAO Cao of 1800 years ago. J Hum Genet, 57(3):216-8.
62. Sykes B, Irven C: (2000)Surname and the Y-chromosome. Am J Hum Genet, 66: 1417-1419.
63. Xue Y, Wang Q, Long Q, et al. (2009) Human Y chromosome base-substitution mutation rate measured by direct sequencing in a deep-rooting pedigree. Curr Biol, 19(17):1453-7.
64. Gusmão L, Sánchez-Diz P, Calafell F, et al. (2005) Mutation rates at Y chromosome specific microsatellites. Hum Mutat, 26:520-528.
65. 1000 Genomes Project Consortium: (2010) A map of human genome variation from population-scale sequencing. Nature, 467:1061-73.
http://blog.sciencenet.cn/blog-348453-699717.html 此文来自科学网王传超博客,转载请注明出处。




